Notice
Recent Posts
Recent Comments
Link
| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | ||
| 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| 13 | 14 | 15 | 16 | 17 | 18 | 19 |
| 20 | 21 | 22 | 23 | 24 | 25 | 26 |
| 27 | 28 | 29 | 30 |
Tags
- 태블로
- 자격증
- tableau
- 이것이 코딩테스트다
- pandas
- Google ML Bootcamp
- 통계
- r
- Python
- scikit learn
- 데이터 분석
- 파이썬
- sklearn
- 데이터 전처리
- matplotlib
- 머신러닝
- SQL
- 딥러닝
- ADsP
- ML
- 회귀분석
- SQLD
- 이코테
- pytorch
- IRIS
- 코딩테스트
- 데이터분석준전문가
- 시각화
- Deep Learning Specialization
- 데이터분석
Archives
- Today
- Total
함께하는 데이터 분석
[EDA] SVD with R 본문
안녕하세요!
오늘은 Singular Value Decomposition의 약자인 SVD에 대해 R을 통해 알아보겠습니다.
우선 코딩에 필요한 파일을 올려놨습니다.
그럼 시작해볼게요!
load("face.rda") #파일 불러오기
image(t(faceData)[, nrow(faceData):1])
svd1$d #singular value
>>> [1] 1.977887e+01 1.513802e+01 1.213935e+01 8.427234e+00 6.200006e+00
[6] 4.936858e+00 4.402278e+00 3.967227e+00 3.743197e+00 3.017167e+00
[11] 2.967196e+00 2.406314e+00 1.899693e+00 1.555837e+00 1.492379e+00
[16] 1.325793e+00 1.275718e+00 1.220815e+00 1.161417e+00 9.251120e-01
[21] 7.397267e-01 6.915633e-01 5.837371e-01 5.438131e-01 4.750496e-01
[26] 4.003466e-01 3.412362e-01 2.582115e-01 1.829855e-01 1.093230e-01
[31] 1.306565e-02 8.755695e-15
plot(svd1$d)
총 32개의 singular value값이 있습니다.
이때 singular value의 제곱 값이 eigen value의 값과 일치합니다.
이제 분산을 살펴보면 아래와 같습니다!
# eigen value값을 총 eigen value로 나눈 누적분포 == 분산
plot(cumsum(svd1$d^2/sum(svd1$d^2)),
pch = 19, xlab = "Singular vector", ylab = "Variance explained")
abline(h=0.8, col='red') #분산의 80퍼 부근 표시
따라서 4개만 있으면 전체 분산의 80% 정도를 표현할 수 있습니다!
그 이상은 분산에 미치는 영향이 미미한 것을 위의 그래프를 통해 알 수 있습니다.
approx1 <- svd1$u[, 1] %*% t(svd1$v[, 1]) * svd1$d[1] #한개로 표현
approx4 <- svd1$u[, 1:4] %*% diag(svd1$d[1:4]) %*% t(svd1$v[, 1:4]) #4개로 표현
approx10 <- svd1$u[, 1:10] %*% diag(svd1$d[1:10]) %*% t(svd1$v[, 1:10]) #10개로 표현par(mfrow = c(1, 4)) #1행 4열로 4개 한번에 보기
image(t(approx1)[, nrow(approx1):1], main = "1 vector")
image(t(approx4)[, nrow(approx4):1], main = "4 vectors")
image(t(approx10)[, nrow(approx10):1], main = "10 vectors")
image(t(faceData)[, nrow(faceData):1], main = "Original data")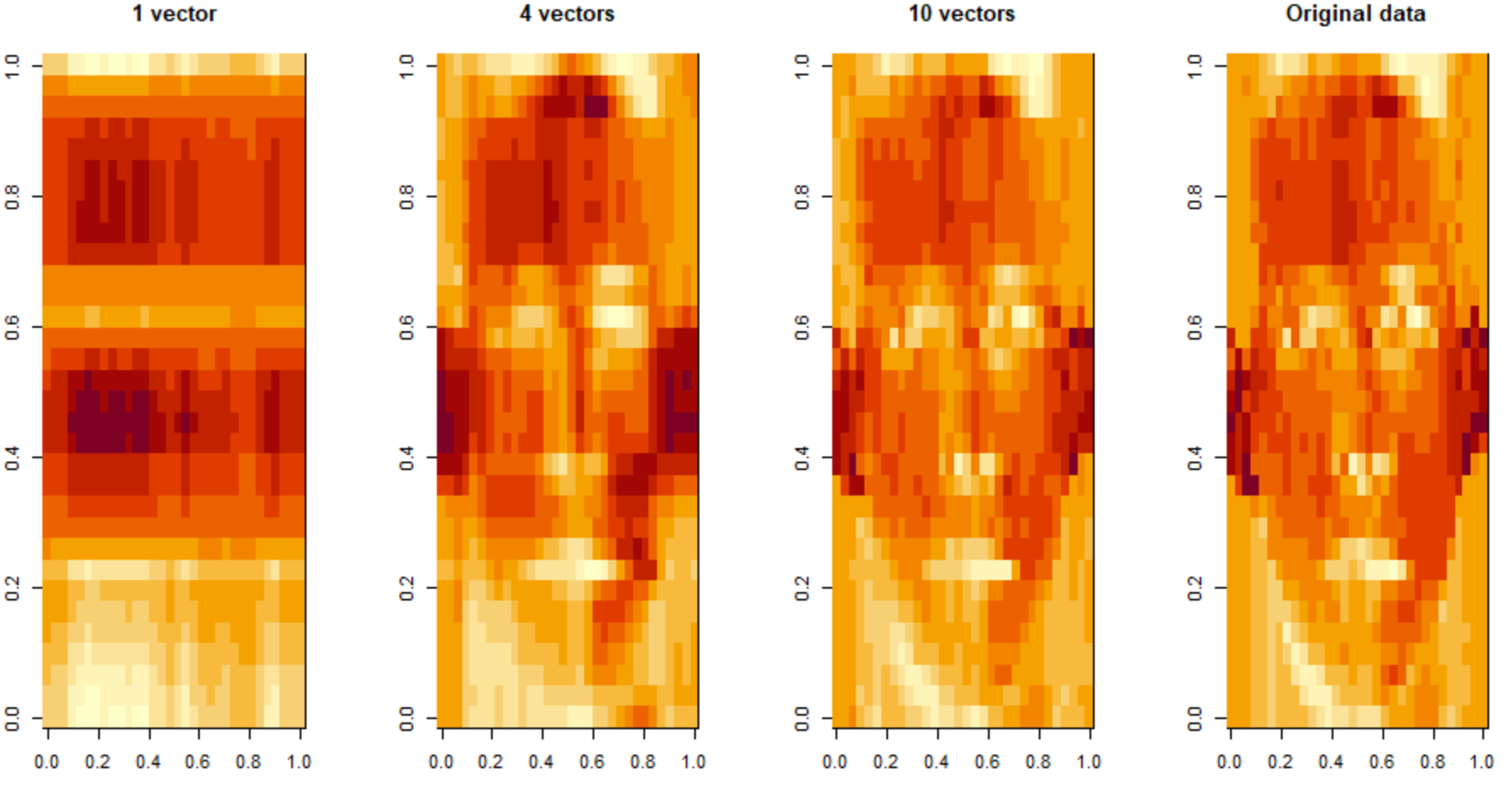
위의 그림을 살펴보면 1개로는 사람인지 인식조차 할 수 없지만
4개는 사람의 형태를 알 수 있는 것을 확인할 수 있습니다.
감사합니다!

'통계학과 수업 기록 > EDA' 카테고리의 다른 글
| [EDA] FA with R (0) | 2022.02.06 |
|---|---|
| [EDA] PCA with R (0) | 2022.02.06 |
| [EDA] K-Means Clustering with R (0) | 2022.02.02 |
| [EDA] Hierarchical Clustering with R (0) | 2022.02.02 |




